
Protenix是字节跳动开源的一款面向蛋白质(及核酸-蛋白复合物)结构预测与相关分析的工具库,基于Apache License 2.0协议开源,代码托管于 [GitHub – bytedance/Protenix],聚焦于解决生物大分子结构预测的高效性、易用性和硬件兼容性问题。
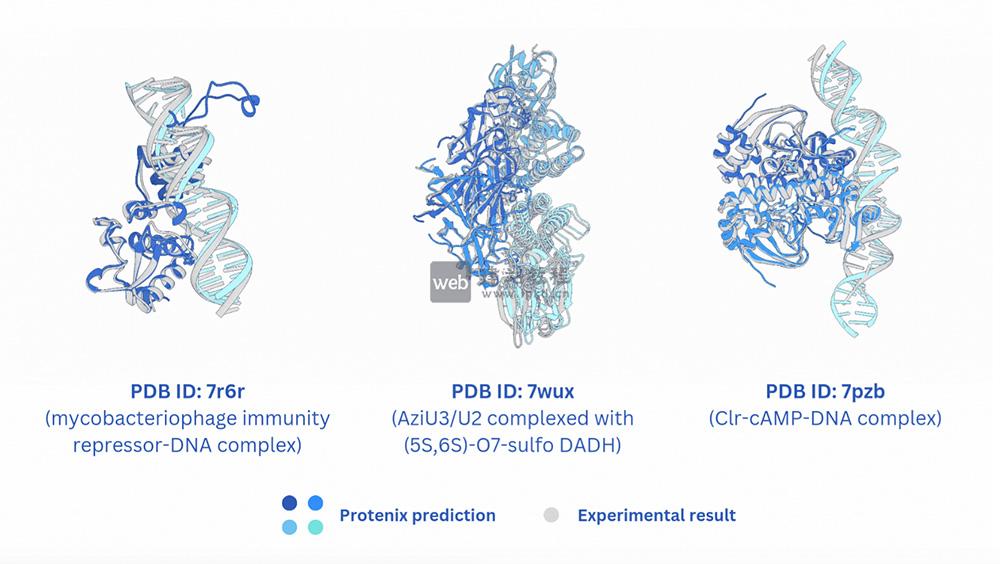
Protenix功能特点:
1、硬件兼容性与高性能:
– 跨硬件适配:针对消费级 GPU(RTX 3090/4090)做了 Triton 内核兼容优化,当 Triton 不可用/不支持时,自动降级为 PyTorch 原生实现,无需修改代码即可在消费级硬件运行;
– 高效计算优化:
– 采用 Triton 编写的自定义注意力内核(`tri_attention`),并通过自动调优(autotune)提升注意力计算效率;
– 实现融合版 AdamW 优化器(Fused AdamW),针对 CUDA 设备加速训练过程;
– 分布式训练支持:集成 PyTorch 分布式(`torch.distributed`),支持多卡训练与 loss 异常检测(NaN/Inf 检查)。
2、多分子类型支持:
– 支持蛋白质链(ProteinChain)、DNA 序列、RNA 序列的结构预测,可定义分子拷贝数(count);
– 支持配体(Ligand)、离子(Ion)建模,支持 CCD 格式的修饰类型(如 CCD_P4G、CCD_MG)与位点指定;
– 提供分子修饰可视化与编辑组件(基于 ipywidgets 的交互式界面),支持手动添加/删除修饰、校验序列合法性(如蛋白序列仅允许 ARNDCQEGHILKMFPSTWYVX 等字符)。
3、易用性与工程化:
– 配置化管理:提供统一的配置加载/解析/保存工具(`protenix.config`),支持训练参数、模型参数的灵活配置;
– 完整的工程链路:
– 内置 CI/CD 流程:GitHub Actions 自动完成代码 lint、测试、PyPI 发布;
– 标准化代码规范:所有核心文件包含字节跳动版权声明与 Apache 2.0 协议头,代码结构清晰(utils/、model/、data/、web_service/ 等模块解耦);
– 交互式工具:提供基于 ipywidgets 的 Web 可视化组件(`protenix.web_service.viewer`),支持序列编辑、修饰添加、共价键定义等可视化操作。
4、可扩展性与开源协作:
– 清晰的贡献流程:提供 CONTRIBUTING.md 规范,支持通过 Issue 提交问题、PR 提交代码,鼓励设计文档(Design Docs)先行的协作模式;
– 模块化设计:核心模块(训练工具、Tokenizer、注意力内核、配置管理)解耦,便于自定义扩展(如新增优化器、自定义注意力内核);
– 开源协议友好:Apache 2.0 协议允许商用、修改、分发,仅需保留版权声明与协议文本。
Protenix快速上手教程:
1、环境准备:
Protenix 基于 Python 3.11 开发,建议使用虚拟环境部署:
# 克隆代码库 git clone https://github.com/bytedance/Protenix.git cd Protenix # 安装依赖 pip install --upgrade pip pip install -r requirements.txt # 可选:安装开发依赖(测试/lint) pip install flake8 pytest
2、基础使用:序列输入与结构预测:
Protenix 支持 JSON 格式的输入文件定义分子序列与修饰,示例参考 examples/example.json:
{
"sequences": [
{
"proteinChain": {
"sequence": "MGSSHHHHHHSSGLVPRGSHMSGKIQHKAVVPAPSRIPLTLSEIEDLRRKGFNQTEIAELYGVTRQAVSWHKKTYGGRLTTRQIVQQNWPWDTRKPHDKSKAFQRLRDHGEYMRVGSFRTMSEDKKKRLLSWWKMLRDNDLVLEFDPSIEPYEGMAGGGFRYVPRDISDDDLLIRVNEHTQLTAEGELLWSWPDDIEELLSEP",
"count": 1,
"msa": {
"precomputed_msa_dir": "./examples/7r6r/msa/1",
"pairing_db": "uniref100"
}
}
},
{
"dnaSequence": {
"sequence": "TTTCGGTGGCTGTCAAGCGGG",
"count": 1
}
}
],
"name": "7r6r"
}
核心步骤::
1、定义蛋白/DNA/RNA 序列、拷贝数、MSA(多序列比对)路径;
2、(可选)添加配体/离子(如 CCD_MG,指定拷贝数);
3、调用模型训练/预测接口,基于配置文件指定训练参数(学习率、优化器、硬件类型等)。
3、模型训练:
Protenix 提供封装好的优化器工具(`protenix.utils.training`),可快速构建训练流程:
import torch
from protenix.utils.training import get_adamw, get_optimizer
from protenix.config import load_config
# 加载配置
config = load_config("path/to/config.yaml")
# 初始化模型(示例)
model = YourProteinModel() # 自定义/加载Protenix内置模型
device = "cuda" if torch.cuda.is_available() else "cpu"
model.to(device)
# 构建优化器
optimizer = get_optimizer(
configs=config,
model=model,
param_names=["backbone", "attention"] # 可选:指定微调参数
)
# 训练循环(示例)
for epoch in range(config.train.epochs):
loss = model.train_step() # 自定义训练步
# 检查loss是否异常
from protenix.utils.training import is_loss_nan_check
if is_loss_nan_check(loss):
print("Loss异常,终止本轮训练")
continue
optimizer.zero_grad()
loss.backward()
optimizer.step()
4、交互式可视化:
Protenix 提供基于 ipywidgets 的交互式组件,可快速编辑分子序列与修饰:
from protenix.web_service.viewer import DnaRnaProteinEntityWidget, LigandIonCCDEntityWidget # 初始化蛋白/DNA/RNA编辑组件 widget = DnaRnaProteinEntityWidget() display(widget) # 在Jupyter Notebook中显示 # 初始化配体/离子编辑组件 ligand_widget = LigandIonCCDEntityWidget() display(ligand_widget) # 获取编辑后的结果(自动校验序列合法性) result = widget.get_result()
Protenix应用场景:
1、基础科研:蛋白质结构预测:
– 针对未知功能的蛋白序列,预测其三维结构,辅助功能注释;
– 研究突变(如氨基酸替换)对蛋白结构的影响,结合修饰位点建模(如磷酸化、配体结合)。
2、药物研发:靶点-配体复合物建模:
– 建模蛋白-小分子配体/离子复合物结构,辅助药物分子设计;
– 分析DNA/RNA-蛋白复合物的相互作用,研究基因调控机制。
3、硬件适配验证:跨平台计算:
– 在消费级GPU(RTX 3090/4090)上验证蛋白结构预测流程,降低科研成本;
– 对比 Triton 加速版与原生 PyTorch 实现的性能差异,优化计算流程。
4、工具链二次开发:
– 基于 Protenix 的 Triton 注意力内核,自定义生物大分子注意力机制;
– 扩展优化器、训练流程,适配特定场景(如低精度训练、超大序列预测);
– 集成到生物信息学平台,提供结构预测的可视化交互功能。
DreamID-Omni虚拟数字人模型,清华 × 字节跳动统一可控以人为中心音视频生成框架
Steerling-8B模型使用入口,80亿参数规模,在1.35万亿Token语料上训练完成
Protenix-v1模型使用入口,开源生物分子结构预测的新标杆
ComfyUI-LaoLi-Shadow安装插件入口,面向ComfyUI的底层性能加速插件
标签: GitHub仓库, Seed团队, 免费开源工具库, 字节跳动
上面是“Protenix官网使用入口,一款面向蛋白质结构预测与相关分析的工具库”的全面内容,想了解更多关于 IT知识 内容,请继续关注web建站教程。
当前网址:https://m.ipkd.cn/webs_30761.html
声明:本站提供的所有资源部分来自互联网,如果有侵犯您的版权或其他权益,请发送到邮箱:admin@ipkd.cn,我们会在看到邮件的第一时间内为您处理!

 javascript关于ajax请求数据的基本用法
javascript关于ajax请求数据的基本用法 OpenCompass:上海AI实验室开源的大模型一站式开放评测体系
OpenCompass:上海AI实验室开源的大模型一站式开放评测体系